|
 |
|
[월간] 과학과 기술 기고문
| ||
| 2021년 07월 |
Focus | mRNA 백신의 화학적 원리와 구조
| |
| 2017년 05월 |
특집 Ⅱ┃유전자가위로 편집하는 DNA 서열 정보 | |
| 2015년 11월 |
Focus┃2015 노벨화학상: 분자수준에서 DNA 손상복구 기작 밝혀 | |
| 2013년 11월 |
Focus┃단일 DNA 분자 손상 부위 형광 표지 방법 개발 | |
| 2012년 10월 |
[특집Ⅰ] 칩위의 실험실, LOC의 세계 - 랩온어칩을 이용한 생체 분자 연구 | |
현대의 광학 현미경 기술이 도래한지 100 여년이 지났지만 형광 표지된 단일 분자 DNA (Single Molecule DNA)를 광학 현미경을 이용하여 최초로 관찰할 수 있게 된 것은 겨우 30여년 밖에 지나지 않았다. 물론 단일 분자 DNA에 대한 분자생물학적 연구는 전자 현미경(Electron microscope)의 발전으로 인하여 이전부터 이뤄지고 있었지만1,2 생물학적인 환경을 관찰하는데 있어서 전자 현미경은 명확한 한계를 지니고 있었다. 가장 큰 제한요소는 전자 현미경에서 관찰하기 위해서는 진공 장치 환경에 맞추어 시료를 건조시켜야 하며 따라서 생물학적인 분자들의 동역학을 분석하는데 한계가 있고 또한 살아있는 분자를 관찰 할 수 없다. 이러한 제한요소를 가진 전자 현미경과는 다르게 1980년대 일본 교토대학의 Mitsuhiro Yanagida (柳田充弘) 교수 연구팀에 의해 발표된 최초의 광학 현미경(Optical microscope)을 통한 단일 분자 DNA의 관찰은 광학 현미경을 통한 생물학적 분자들의 연구에 있어 새로운 시각을 가져왔으며 이후에 생물학적 단일 분자의 새로운 분석 방법들이 개발되는 촉매의 역할을 하였다.3 1989년 Nature와 Science에는 DNA 분리에 이용되는 전기영동에서 실제로 DNA 분자가 어떻게 움직이는 지를 단일 분자의 영상화하여 발표되었으며 이를 통하여 분자 동역학 연구의 새로운 장이 열리게 되었다 [그림 1].4,5 단일 분자 DNA에 대한 연구는 그 이후로도 주목할 만한 많은 발전을 이룩하였다. 특히 단일 분자 DNA 분석을 광학 현미경으로 할 수 있게 된 것은 형광 염료의 개발과 밀접한 관련이 있다. TOTO 시리즈라고 불리는 삽입 염료(intercalating dye)의 개발이 중요한 기폭제가 되었는데 이중나선의 DNA 사이에 염료 분자가 끼어 들어간 경우에만 형광을 내기 때문에 매우 선명하게 단일 분자 DNA를 영상화 할 수 있는 장점을 가지고 되었다. 본 총설에서는 지금까지 연구되어 온 단일 분자 DNA 영상화 (Single Molecule DNA Imaging) 를 이용한 생물-물리학적 연구와 이를 통해서 유전정보에 분석하는 방법들에 대한 연구를 소개하고자 한다.
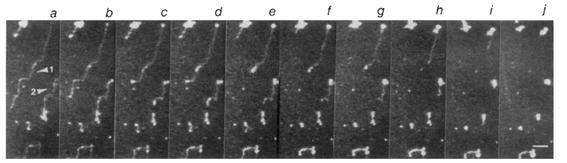
[그림 1] 아가로스 겔(Agarose gel)내부에서의 박테리아 DNA의 신장과 이완4
1. 단일분자 DNA의 고정과 조작
단일 분자를 이용한 연구에 관심이 높아지면서 단일 분자를 고정하고 조작할 수 있는 장비들 또한 빠르게 발전 되어왔다. [그림 1]에서 소개한 방법과 같이 겔 내부에서 DNA분자를 길게 펴는 연구로부터 단일 분자 DNA 의 연구가 시작되었지만 이 방법에서는 분자가 고정되어 있지 않기 때문에 분자를 마음대로 조작하는 것이 쉽지 않았다. 1990년대부터 지난 20년간 어떻게 효과적으로 DNA 분자 하나하나를 자유롭게 조작할 수 있도록 할 것인가에 대한 여러 가지 방법들이 개발되어 왔는데 이에 대한 소개를 하고자 한다.
1) 광핀셋 (Optical Tweezer)을 이용한 단일분자 DNA의 연구
단일 분자 DNA를 고정하는 방법 중에서 주목할 만한 기술 중 하나는 광핀셋 기술을 이용하는 방법이다. 광핀셋은 1970년대에 벨연구소에 있던 Ashkin 박사에 의해서 처음 개발되었다. 이 방법은 작은 물체에 강한 빛을 받으면 운동량 보존 법칙에 따라 물체가 움직이게 된다는 원리를 기초로 하고 있는데 이 원리와 광학장치를 적절히 이용하면 미세한 입자를 원하는 위치에 고정할 수 있고 또한 자유롭게 위치를 조절할 수도 있다. 1991년 University of Oregon의 Bustamante 교수 (현 UC Berkeley)는 이 방법을 응용해서 DNA 분자 하나를 고정하는 방법을 개발하였다.6 미세입자에 DNA 분자를 연결한 다음에 광핀셋 기술을 이용하여 미세입자를 조작하게 되면 단 하나의 DNA 분자를 잡아 당겨서 길게 늘어뜨릴 수도 있고 DNA 분자에 걸리는 힘도 측정할 수 있게 된다. 이 방법은 곧이어 스탠포드 대학의 Steven Chu 교수 그룹에서 보다 본격적으로 연구되어 졌으며 약 10여 편의 논문을 Science에 시리즈로 소개 하였는데 [그림 2]는 Steven Chu 그룹에서 1994년에 Science에 발표한 논문이다.
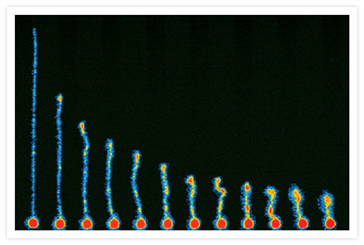
[그림 2] 광핀셋에 의해 고정된 DNA의 신장과 완화 현상.7
[그림 2]에서보면 단일분자 DNA를 미세 입자에 붙여준 다음 광핀셋을 이용하여 미세입자의 위치를 고정하고 주변에 유체의 흐름을 만들어 주면 DNA는 길게 늘어나게 된다. 그리고 주변 유체의 흐름을 멈추었을 때 신장 되었던 단일 분자 DNA가 시간에 따라 점점 말려서 길이가 짧아지는 이완 현상을 영상으로 보여주고 있다. 이 연구는 고분자 물리학에 매우 중요한 발전을 가져 왔는데 DNA와 같은 고분자가 힘에 의해서 늘어났다가 엔트로피 효과 때문에 다시 줄어드는 현상은 1950년대부터 고분자 물리학 이론을 연구하는 물리학자들에 의해서 연구되어져 왔지만 이를 실험적으로 구현하는 것이 상당히 어려웠다. 그런데 [그림 2]의 실험을 통해서 고분자 물리학의 이론들을 매우 명확하게 연구할 수 있게 되면서 고분자 물리학이 비약적으로 발전할 수 있는 계기를 마련하였다.
광핀셋을 이용하는 단일 분자 DNA 실험이 고분자의 물리학적 특성을 연구하는데 매우 유용한 도구로 널리 이용되어 왔지만 이와 같은 연구 방법은 유전학 분야로 넘어가는 것은 몇 가지 단점 때문에 쉽지 않았다. 첫째로 광핀셋 시스템을 구성하는 것이 간단하지 않다. 광학을 전공하는 물리학자들도 이를 구성 할 때 많은 비용과 긴 시간이 걸린다. 최근에는 사용하기 편리하도록 만들어진 상용패키지가 출시되었으나 아직 많이 보급되지 못한 형편이다. 또한 값비싼 레이저 한 대를 이용하여 분자 하나 밖에 분석할 수 없다는 것도 광핀셋을 생물학적 응용에 적용하는데 큰 단점이다. 물리학 이론을 검증하는 수준의 연구에서는 대개 문제가 되지 않지만 유전자 정보를 얻는 생물학적 응용에서는 수백만 개의 분석을 동시에 수행할 수 있는 능력이 매우 중요한데 광핀셋을 이용하는 방법으로는 이러한 연구를 하는 것이 거의 불가능하다고 할 수 있다. 따라서 최근에는 단일 분자 DNA를 연구하는 분야에서 광핀셋을 이용하는 연구보다는 수많은 분자를 병렬적으로 동시에 분석할 수 있는 방법의 개발이 주목을 받고 있다.
2) 개질된 표면(Modified surface)을 이용한 DNA 분자의 병렬 고정화
수많은 단일 분자 DNA를 병렬로 표면에 고정하는 방법은 1994년 프랑스의 파스테르 연구소의 Bensimon 박사에 의해서 처음으로 개발되어 분자 빗질 (Molecular Combing)이라는 이름으로 소개되었다.8 분자 빗질에서는 DNA가 들어 있는 용액에 유리를 넣었다가 로봇 팔을 이용하여 DNA 분자가 끊어지지 않도록 천천히 유리를 끌어 올리면 유리 표면에 수많은 DNA 분자가 길게 펴지는 현상을 발견할 수 있었다. 이 방법은 수많은 DNA 분자를 병렬로 고정할 수 있는 방법으로 fiber FISH (Fluorescent in situ hybridization)을 할 수 있는 방법으로 주목을 받았지만 몇 가지 문제점들을 극복하기 힘들었기 때문에 지금은 많이 쓰이지 않고 있다. 가장 큰 문제점은 이 방법으로 얻어진 DNA 분자는 너무 길게 늘어나서 대개는 150% 이상으로 늘어나게 되고 그러면 더 이상 생화학적 처리를 하는 것이 불가능 하다는 것이었다.
비슷한 시기인 1995년 뉴욕대학의 Schwartz 교수 (현 UW-Madison) 그룹은 단일 분자 DNA를 양전하로 개질된 표면에 고정하는 방법을 발표하였다.9 Schwartz 교수 그룹은 앞서 아가로스 겔을 이용하여 DNA를 고정하고 제한효소로 처리하여 유전자 지도를 만드는 방법인 시각적 지도 작성법 (Optical Mapping) 방법을 개발하였었는데 아가로스 겔에서는 DNA 분자가 시간이 지남에 따라 계속 점점 작아지는 문제점이 있었다.10 이 문제를 해결하기 위해서 양전하로 개질된 표면을 개발하였고 이 표면에 결합된 DNA 분자는 시간이 지나도 변형되지 않는 다는 것을 알 수 있었다. 일반적으로 DNA 분자는 인산기 때문에 음이온을 가지고 있어서 양이온을 가진 개질된 유리표면에 정전기적인 인력에 의해서 고정할 수 있으며 이 방법으로 고정된 DNA는 제한 효소 등의 생화학적 처리를 하는 데에 전혀 문제가 없다. 이 방법은 후에 마이크로 유체 장치와 결합하여 발전하였는데 마이크로 채널로 방향이 정해지면 수많은 DNA 분자가 한 방향으로 길게 늘어나서 바닥에 고정된다.11 이 방법은 지금까지 수많은 유전체 사업에서 DNA 서열을 맞추는 데 기준이 되는 유전자 지도 방법으로 발전되어 왔으며 2010년에는 이 방법을 이용한 인간 유전체 지도를 만드는 데에 성공하였다[그림 5A].12
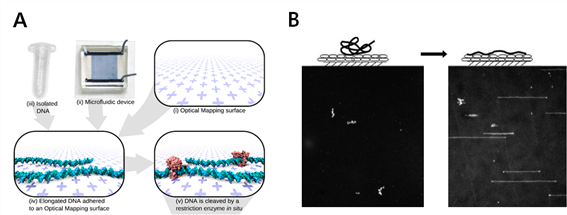
[그림 5] (A) 양이온을 가진 화합물로 전하를 띈 유리표면을 만들어 단일 분자 유전체를 분석하는 방법12, (B) 단백질로 처리된 표면을 이용하여 단일 분자 DNA의 고정.13
하지만 양전하 표면은 DNA 분자 뿐 아니라 음전하를 많이 가지고 있는 단백질, RNA, 핵산 등의 수많은 다른 생화학적 물질들도 흡착하는 문제점을 가지고 있기 때문에 다양한 목적으로 응용하는 데에 문제점이 있다. 이러한 무작위적 흡착을 극복하기 위해서 최근에 필자의 연구실에서는 DNA 분자에 흡착 성질을 가지는 아비딘 (Avidin) 단백질을 찾아내고 이를 표면에 처리함으로써 다른 생화학적 물질은 흡착되지 않고 선택적으로 DNA 만을 흡착할 수 있는 새로운 시스템을 개발하였다. 특히 이 방법은 pH에 변화와 금속이온의 농도에 따라 탈부착이 가능하므로 기존의 전하를 가진 표면에 비해 더 많은 응용이 가능할 것으로 생각된다. [그림 5B]
위와 같은 DNA를 표면에 고정화 하는 방법들이 중요한 이유는 단일 분자 DNA로부터 유전정보를 얻기 위해서는 DNA를 길게 펴 주는 것이 매우 중요하다. 위에서 설명한 두 가지의 실험 방법들은 DNA를 표면에 고정시킴으로서 제한 효소에 의한 DNA의 절단이나 DNA 형광표지에 의하여 이후에 설명할 시각적 지도 작성법 사용되며 DNA의 방향성을 결정해 주기 때문에 유전정보를 순서대로 읽을 수 있도록 해준다.
3) 한쪽 끝을 고정하고 단일 분자 DNA를 길게 펴는 방법
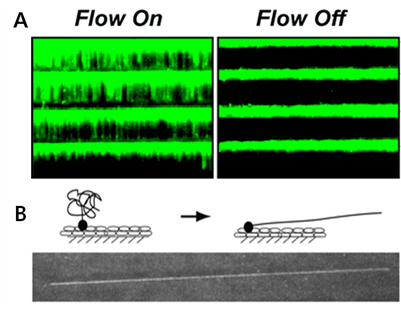
[그림 4] (A) 지질 이중층을 이용한 여러 개의 단일 DNA를 분자의 신장시킨 이미지14,15 (B) 아비딘으로 처리된 표면에 바이오틴을 이용하여 단일 분자 DNA의 한쪽 끝을 결합시킨 이미지.13
한쪽 끝만 고정시키고 DNA를 길게 펴는 새로운 방법으로 2008년 미국 콜로비아 대학의 Greene 교수 연구팀에 의해 개발된 DNA 커튼이라는 방법이 있다[그림 4A].14 이 방법에서는 지질층에 아비딘과 한쪽 끝이 바이오틴으로 표지된 된 DNA를 넣어주어 지질 이중층 표면 위를 아비딘이 결합된 DNA가 흐를 수 있게 하였다. 그리고 표면에 식각된 구조물이나 크롬층을 만들어 주고 일정한 방향으로 흐름을 발생시킴으로써 DNA 분자들의 길게 펴지도록 하였고 그 DNA 위에서 단백질 결합하는 위치를 분석할 수 있었다. 이를 응용하여 2011년에는 필자의 연구 그룹에서 유사한 방법을 개발하였는데 아비딘을 유리 표면에 고정화 시키고 그 위에 동일하게 바이오틴 표지된 DNA를 흘려주어 한쪽으로 유체의 흐름을 주어 DNA 분자를 길게 펼 수 있었다. [그림 4B]에서 보면 DNA 분자의 한쪽 말단을 고정하고 유체의 흐름을 이용하여 100%에 가깝게 펴진 DNA 분자를 관찰할 수 있었다. 이는 앞에서 설명한 광핀셋을 이용한 유전체 고정화 방법과 유사한 방법으로서 유전체의 한쪽 끝에 바이오틴을 가진 올리고머를 리가제로 결합시켜 DNA의 한쪽 끝에 바이오틴을 표지화한 이후에 바이오틴과 결합력을 가지는 다른 물질들을 통해서 한쪽을 표면에 고정화 시키는 방법과 유사하지만 하나의 분자를 분석하는 대신 여러 개의 분자들을 동시에 분석할 수 있다는 장점을 가지고 있다.
4) 나노 구조물(Nano structure)을 이용한 단일분자 DNA 조작
리소그래피(Lithography) 기술의 발전은 미세한 나노 스케일의 구조물을 쉽게 만들 수 있도록 해 주었으며 나노 구조물의 발전은 단일 분자 DNA의 분석 방법의 개발에 영향을 주었다. 나노 구조물 들을 이용한 단일 DNA의 분석 방법은 두 가지로 나누어 볼 수 있는데 첫째 아가로스(Agarose)와 같은 기둥이나 올가미 형태의 나노 구조물을 생성하여 전기장을 하여 분석 하는 방법이 있으며 여러 그룹에서 연구되고 있다[그림 6].16
인공 겔 (Artificial Gel)이라고 불리는 나노 구조물의 특징은 단일 분자 수준에서 전기영동장치와 같은 DNA의 사이즈 별 분리가 가능하며 분리되는 과정을 이미징 함으로써 전기 영동장치의 DNA 분석 과정을 좀 더 세밀하게 알 수 있도록 해준다.

[그림 6] (A) Lithography를 이용한 나노사이즈 기둥들. (B) 나노 기둥들을 통한 단일 분자 DNA의 이동.16
그런데 인공 겔을 이용한 전기영동을 구현하는 대신에 나노 채널 형태의 구조물을 만들어 이 안에 단일 분자의 DNA를 넣어 주게 되면 DNA 분자는 좁은 공간 때문에 길게 펴지게 되는데 이 방법을 이용하면 분리를 하지 않고 또 화학적인 고정을 하지 않고 길게 펴진 DNA를 고정할 수 있는 방법이 된다. [그림 7]
나노 미터 크기의 채널에 고정화된 단일 분자 DNA는 채널의 크기(Dimension)와 이온세기에 의해 채널 내부에서 길어지는 정도가 다르며 이를 이용하여 단일 사슬 형태의 DNA 분자의 물리적 특성에 대한 연구 뿐 만 아니라 DNA의 유전적 정보를 얻을 수 있다.
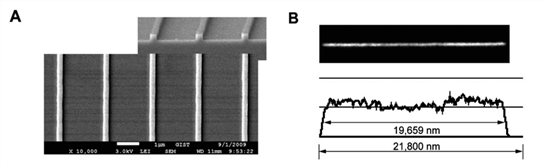
[그림 7] (A) 단일 분자 DNA 분석에 사용되는 나노 채널의 전자현미경 이미지 (B) 나노 채널에 의해 90%까지 길게 늘어진 DNA 단일 분자의 길이 분석.17
2. 단일 분자 DNA를 이용한 유전정보 분석 방법
앞서 단일 분자 DNA의 고정과 조작할 수 있는 방법들에 대하여 알아보았다 이러한 방법들은 긴 DNA 분자의 물리적인 성질을 연구하는데 사용되며 또한 DNA의 염기서열 정보를 얻기 위해서도 사용된다. 시각적 지도 작성법, 나노 유체 장치를 이용한 단일 DNA 분석방법과 같은 단일 분자를 이용하여 유전적 정보를 얻어 내는 방법들에 대하여 살펴보고 단일 분자를 이용한 방법들이 가지는 장점과 새로운 연구 사례에 대하여 알아보고자 한다.
1) 제한 효소(Restriction enzyme)를 이용한 시각적 지도 작성법
1993년 Science에 Schwartz 교수 그룹은 제한 효소(Restriction enzyme)를 이용한 시각적 지도 작성법(Optical mapping)을 발표하였다. 시각적 지도 작성법에서는 제한 효소를 이용하면 효소에 따라 특정 염기서열을 인식하여 절단해 주는데 이를 이용하면 앞에서 설명했던 양전하를 띈 유리 표면에 마이크로 유체 장치를 이용하여 붙여놓은 DNA의 특정 염기 서열들을 잘라 줌으로써 바코드 형태의 유전자 지도를 만들게 된다.
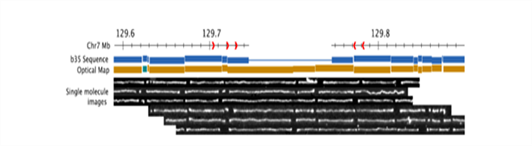
[그림 8] 시각적 지도 작성법으로 인간 유전체를 분석12
이 방법을 발전 시켜서 2010년에는 시각적 지도 작성법으로 4명의 인간 유전자를 분석하였으며, 수십 kb에서 수 Mb 크기의 긴 DNA에 발생한 유전자 다형성을 발견하였다[그림 8]. 이를 통해 단일 분자를 이용한 유전자 분석법인 시각적 지도 작성법이 긴 유전체의 변형 즉 삽입, 결손, 전좌, 반복 등을 분석하였다.
2) 나노유체장치(Nanofluidic device)를 이용한 단일분자 DNA 분석.
앞서 나노채널 장치에 대하여 설명하였듯이, 나노채널의 크기가 DNA 분자의 지지길이 (persistance length:~50nm)에 이르면 채널 내부의 DNA 들은 채널을 따라서 길게 신장하게 되고 따라서 나노 채널을 이용하면 단일 분자 DNA의 염기서열 분석 할 수 있다. 나노 채널의 경우에는 채널의 크기를 이용하여 DNA를 손쉽게 길게 펼 수 있다는 장점을 가지고 있지만 앞서 설명하였던 제한 효소를 사용한 지도 작성법은 문제점이 있는데 DNA가 고정되어 있지 않기 때문에 잘려진 DNA가 자유롭게 움직여서 바코드와 같은 패턴을 만들어 낼 수가 없다. 이러한 문제를 해결하기 위해서 2007년 두 편의 논문이 서로 다른 그룹에서 발표되었는데 그 원리는 비슷하다. 이를 살펴보면 특정한 서열 위치에 틈새 번역 (Nick translation)을 이용하여 단일 분자 DNA에 형광표지를 입혀줌으로써 특정 염기서열의 존재 여부를 단일 분자 수준에서 볼 수 있는 방법을 개발 하였다[그림 9].18,19
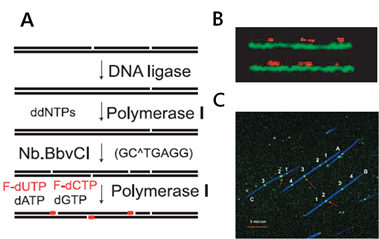
[그림 9] (A) 단일 분자 DNA를 이용한 틈새 번역(Nick translation)방법에 대한 도식18, (B) 나노채널에서의 틈새번역된 DNA 분자18, (C) 틈새 번역후 표면에 고정하고 전반사 형광법 (TIRF)로 분석19
틈새 번역 (Nick translation) 방법은 먼저 틈새 효소 (nicking enzyme)를 사용하는데 이는 제한효소처럼 DNA의 특정 염기서열을 인식하여 잘라주는 역할을 하는데 이때 제한효소와는 다르게 이중나선의 한쪽 나선만 자름으로서 틈새를 만들어준다. 이 만들어진 틈새를 DNA 중합효소를 이용하여 채워주는데 형광물질을 가지고 있는 modified된 dNTP를 사용하여줌으로서 유전체 위에 형광 표지가 될 수 있도록 하는 것이다. 틈새 번역된 DNA 분자 들을 나노채널에 넣어주어 길게 펴면 형광물질로 표지된 부분을 통하여 시각적 DNA 지도 작성법과 같이 유전정보를 얻을 수 있게 된다.
3) DNA 결합 단백질(DNA-binding protein)을 이용한 시각적 지도 작성법의 응용
단일 분자 DNA를 응용한 방법의 하나로써 DNA 결합 단백질을 이용한 지도 작성법은 2009년 UCLA의 Simon Weiss 교수 그룹에서 시작되었다. 형광 표지된 RNA 중합효소를 이용하여 T7 박테리아파지의 17개의 전사인자지도를 단일 분자 DNA 수준에서 만들었다.20
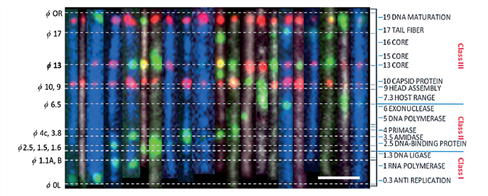
[그림 10] 단일 분자DNA에 단백질을 이용한 참조 표시된(SMILing DNA)와 그에 결합한 RNA 중합효소의 결합 부위지도21
2012년 동일 그룹에서 발표한 논문으로 SMILing DNA(Sequence-specific methyl transferase induced labeling of DNA)를 기준으로 하고 형광 표지를 Methyl transferase를 이용하여 만들어 주었으며, 이를 이용하여 기존의 RNA 중합효소의 결합위치에 기준 지점을 제시 하여 줌으로써 더 정밀한 유전자 지도를 만들 수 있도록 하였다[그림 10].21
4) DNA Fluorocode : 나노미터 분해능을 가진 광학적 유전자 분석법.
빛의 회절 한계는 유전자의 시각적 지도 작성법의 가장 커다란 한계일 것이다. 단일 분자 이미징에 사용되는 빛에 의한 한계는 일반적으로 250-300 nm에 해당하며 이는 DNA의 경우 약 750 bp정도의 낮은 분해능을 가질 수밖에 없다. 이는 광학적 지도 작성법이 가지는 매우 큰 문제이며 이를 해결하기 위하여 벨기에의 Johan Hofkens 교수 그룹은 Fluorocode라는 새로운 방법을 개발한다. Fluorocode는 M.HhaI 이라는 5‘GCGC-3’의 서열을 인지하는 Methyltransferase를 이용하여 단일 분자의 DNA에 형광 염료를 표지한 후에 폴리머 처리된 표면에 고정 시켜 분석한다. 이후에 기존의 방법과는 다르게 높은 분해능을 가지게 하기 위하여 형광물질의 발색 (photobleaching) 현상을 이용하는데 형광 표지된 각각의 염료 들은 레이저의 세기와 시간에 따라 발색되며 이를 시간에 따라 지속적으로 측정하여 PSF(Point Spread Function)을 이용하여 분석하게 되면 약 20 bp정도의 높은 분해능을 가지는 광학적 유전자 지도를 얻을 수 있게 된다 [그림 11].22

[그림 11] Fluorocode에 의해 분석된 20개의 단일 분자 λ DNA, (A) 실험 데이터 (B) 참조 데이터22
지금까지 단일 분자 DNA를 길게 펴서 조작하는 방법과 길게 펴진 DNA에서 유전 정보를 얻어내는 방법에 대해서 살펴보았다. 최근에 주목받고 있는 차세대 서열 분석법은 매우 유용한 방법임에도 불구하고 짧은 염기서열 밖에는 읽어 낼 수 없다는 단점을 가지고 있다. 또한 지난 몇 년 동안 가격이 많이 내렸음에도 불구하고 아직까지는 가격이 너무 비싸다. 이에 비하여 수십 kb에서 수 Mb의 이르는 거대한 DNA를 분자를 이용하는 분석 방법은 차세대 서열 분석법의 단점들을 보완하여 매우 유용한 유전 정보를 제공할 수 있는 새로운 플랫폼이라 할 수 있으며 서열 분석의 가격을 낮추는 데에도 매우 중요한 역할을 할 것으로 기대된다. 또한 분자를 마음대로 조절하고 분자 위에서 일어나는 현상을 직접 관찰할 때에는 물리, 화학, 생물학의 관념적 경계가 사라지게 되며 융합한 학문의 관점에서 자연 현상을 관찰 할 수 있게 만들어 준다. 따라서 이러한 새로운 현상의 관찰은 새로운 학문적 진보의 초석이 될 것으로 기대된다.
참고문헌
1. Price, W. C.; Williams, R. C.; Wyckoff, R. W., Science 1945, 102, 277-278.
2. Herzberg K.; Kleinschmidt, A.; Zentalbl Bakteriol Orig 1959, 174, 1-11.
3. Morikawa, K.; Yanagida, M, J Biochem 1981, 89(2), 693-696.
4. Schwartz, D. C.; Koval, M., Nature 1989, 338(6215), 520-2.
5. Smith, S. B.; Aldridge, P. K.; Callis, J. B., Science 1989, 243(4888), 203-6.
6. Smith, S. B.; Finzi, L.; Bustamante, C.; Science, 1992, 1122-1126.
7. Perkins, T. T.; Quake, S. R.; Smith, D. E.; Chu, S., Science 1994, 264(5160), 822-826.
8. Bensimon, A.; Simon, A.; Chiffaudel, A.; Croquette, V.; Heslot, F.; Bensimon, D.; Science, 1994, 265, 2096-2098.
9. Cai, W. W.; Aburatani, H.; Stanton, V. P.; Housman, D. E.; Wang, Y. K.; Schwartz, D. C., PNatlAcadSciUSA 1995, 92(11), 5164-5168.
10. Schwartz, D. C.; Li, X.; Hernandez, L. I.; Ramanarain, S. P.; Huff, E. J.; Wang, Y. K., Science, 1993, 262, 110-114.
11. Dimalanta, E. T.; Lim, A.; Runnheim, R.; Lamers, C.; Churas, C.; Forrest, D. K.; de Pablo, J. J.; Graham, M. D.; Coppersmith, S. N.; Goldstein, S.; Schwartz, D. C., Anal Chem, 2004, 76, 5293-5301.
12. Teague, B.; Waterman, M. S.; Goldstein, S.; Potamousis, K.; Zhou, S. G.; Reslewic, S.; Sarkar, D.; Valouev, A.; Churas, C.; Kidd, J. M.; Kohn, S.; Runnheim, R.; Lamers, C.; Forrest, D.; Newton, M. A.; Eichler, E. E.; Kent-First, M.; Surti, U.; Livny, M.; Schwartz, D. C., PNatl Acad Sci USA 2010, 107(24), 10848-10853.
13. Kim, Y.; Jo, K., Chem Commun 2011, 47(22), 6248-6250.
14. Fazio, T.; Visnapuu, M. L.; Wind, S.; Greene, E. C., Langmuir 2008, 24(18), 10524-10531.
15. Greene, E. C.; Wind, S.; Fazio, T.; Gorman, J.; Visnapuu, M. L., Method Enzymol 2010, 472, 293-315.
16. Kaji, N.; Tezuka, Y.; Takamura, Y.; Ueda, M.; Nishimoto, T.; Nakanishi, H.; Horiike, Y.; Baba, Y., Anal Chem 2004, 76(1), 15-22.
17. Kim, Y.; Kim, K. S.; Kounovsky, K. L.; Chang, R.; Jung, G. Y.; dePablo, J. J.; Jo, K.; Schwartz, D. C., Lab Chip 2011, 11(10), 1721-1729.
18. Jo, K.; Dhingra, D. M.; Odijk, T.; de Pablo, J. J.; Graham, M. D.; Runnheim, R.; Forrest, D.; Schwartz, D. C., PNatl Acad Sci USA 2007, 104(8), 2673-2678.
19. Xiao, M.; Phong, A.; Ha, C.; Chan, T. F.; Cai, D.; Leung, L.; Wan, E.; Kistler, A. L.; DeRisi, J. L.; Selvin, P. R.; Kwok, P. Y., Nucleic Acid Research , 2007, 35, e16.
20. Ebenstein, Y.; Gassman, N.; Kim, S.; Antelman, J.; Kim, Y.; Ho, S.; Samuel, R.; Michalet, X.; Weiss, S., Nano Lett, 2009, 9, 1598-1603
21. Kim, S.; Gottfried, A.; Lin, R. R.; Dertinger, T.; Kim, A. S.; Chung, S.; Colyer, R. A.; Weinhold, E.; Weiss, S.; Ebenstein, Y., Angew Chem Int Edit 2012, 51(15), 3578-3581.
22. Neely, R. K.; Dedecker, P.; Hotta, J. I.; Urbanaviciute, G.; Klimasauskas, S.; Hofkens, J., Chem Sci 2010, 1(4), 453-460.